В качестве бонуса и завершающего урока я хочу рассказать об ещё одной библиотеке для работы с медицинскими изображениями. А именно о fastai.medical.imaging
Эта библиотеке здорово упрощает работу с DICOM файлами. Давайте посмотрим на нашем уже привычном примере, что она умеет.
Для начала установим fastai. fastai - это библиотека глубокого обучения, которая позволяет быстро и легко обучать нейронные сети.
pip install fastai
pip install kornia
fastai достаточно активно развивается и часто нудно изрядно повозиться с обновлениями и совместимостью версий. Поэтому если вы хотите рекомендую опробовать то рекомендую попробовать его в google colab.
[ -e /content ] && pip install -Uqq fastai
pip install kornia
Импортируем всё необходимое
from fastai.basics import *
from fastai.vision.all import *
from fastai.data.transforms import *
from fastai.medical.imaging import *
import pydicom,kornia,skimage
Тестировать будем всё на том же файле.
dimg = pydicom.dcmread('ID_01fe90211.dcm')
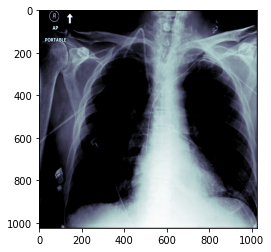
И первая крутая возможность это преобразовать pixel_array в TensorImage типо torch.uint8. А визуализировать можно с помощью метода .show()
ten_img = TensorDicom(dimg.pixel_array)
print(ten_img)
ten_img.show()
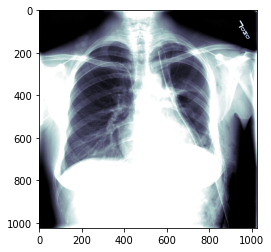
Также можно изменить cmap на bone, который лучше распознает различные области изображения.
class TensorDicom(TensorImage): _show_args = {'cmap':'bone'}
ten_img2 = TensorDicom(dimg.pixel_array)
ten_img2.show()
Вы можете применить windowing двумя способами: указав значения W и L или воспользоваться уже готовыми окнами, например dicom_windows.lungs . Результат будет одинаковый.
plt.imshow(dimg.windowed(w=1500, l=-600), cmap=plt.cm.bone)
plt.imshow(dimg.windowed(*dicom_windows.lungs), cmap=plt.cm.bone)
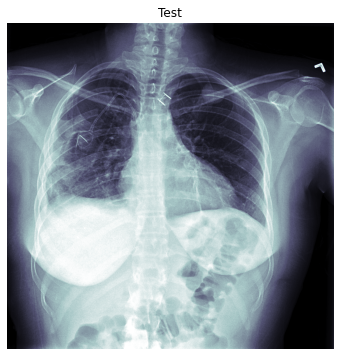
Так же с помощью show вы можете кастомизировать отображение DICOM изображений.
dimg.show(scale=True, cmap=plt.cm.bone, min_px=-1100, max_px=None, ax=None, figsize=(6,6), title='Test', ctx=None)
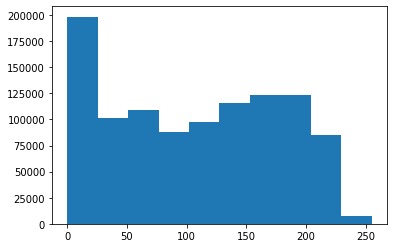
Мы можем просмотреть распределение пикселей DICOM изображения. Гистограмма ниже отображает текущее распределение пикселей, которое показывает диапазон пикселей от 0 до 250.
plt.hist(dimg.pixels.flatten().numpy())
Еще один крутой функционал это масштабирование пикселей. Можно масштабировать по значениям min_px и max_px для пикселей DICOM изображения или указать их вручную.
data_scaled = dimg.hist_scaled()
plt.imshow(data_scaled, cmap=plt.cm.bone)
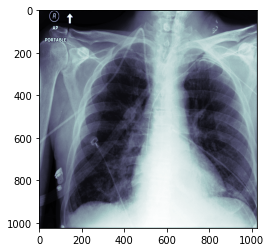
data_scaled = dimg.hist_scaled(min_px=100, max_px=1000)
plt.imshow(data_scaled, cmap=plt.cm.bone);