Как я уже говорил в файлах DICOM помимо изображения содержатся метаданные. Например: возраст, пол, модальность исследования, ФИО, часть тела и положение тела.
Давайте попробуем исследовать наши метаданные. Начнём с того, что мы уже знаем, с чтениям файла
example = 'stage_2_images/ID_01fe90211.dcm'
imagedata= pydicom.dcmread(example)
Извлечем ID пациента, возраст, пол, модальность, часть тела и рентгенологический вид изображения относительно ориентации объекта.
print("ID:", imagedata.PatientID)
print("Возраст:", imagedata.PatientAge)
print("Пол:", imagedata.PatientSex)
print("Модальность:", imagedata.Modality)
print("Часть тела:", imagedata.BodyPartExamined)
print("Рентгенологический вид изображения относительно ориентации объекта:", imagedata.ViewPosition)
Результат будет следующим:
ID :
Возраст : 26
Пол : F
Модальность : CR
Часть тела : CHEST
Рентгенологический вид изображения относительно ориентации объекта изображения : PA
Думаю с ID, возрастом и полом (F-Female, M-Male) всё понятно. Разберемся с модальностью. Основные типы (модальности) поддерживаемых стандартом DICOM медицинских изображений представлены ниже:
И многие другие типы исследований.
Часть тела: CHEST - грудная клетка. С полным списком всех аббревиатур частей тела можно ознакомиться тут. Что касается вида изображения относительно ориентации объекта изображения, то для грудной клетки оно может принимать значения соответствующие прямой (передняя и задняя) и боковой (левая или правая) проекции. PA - передняя, AP - задняя.
Еще отдельно мы можем проверить есть ли в DICOM файле изображение и её размер.
if 'PixelData' in imagedata:
rows = int(imagedata.Rows)
cols = int(imagedata.Columns)
print("Размер изображения : {rows:d} x {cols:d}, {size:d} байт".format(
rows=rows, cols=cols, size=len(imagedata.PixelData)))
Результат - Размер изображения : 1024 x 1024, 158236 байт
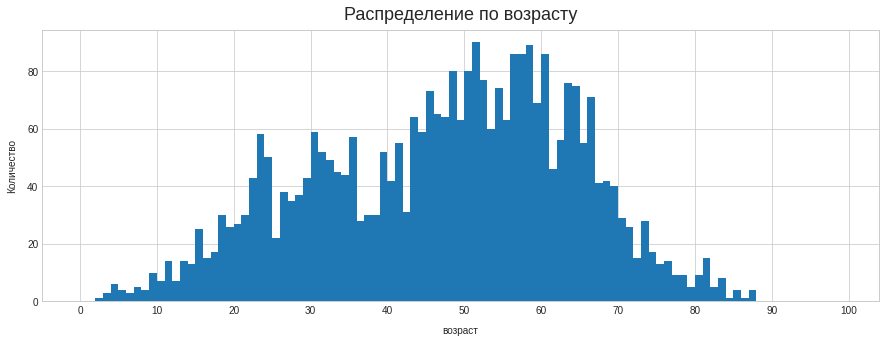
А теперь давайте сделаем небольшой обзор данных. Например, посмотрим какое распределения возраста и пола в нашем датасете.
Начнём с распределения возраста. Построим гистограмму.
from glob import glob
import pandas as pd
import seaborn as sns
datalist = sorted(glob("stage_2_images/*.dcm")) # создадим лист содержащий пути ко всем файлам
# Для удобства анализа создадим датафрейм с метаданными
patients = []
for t in datalist:
data = pydicom.dcmread(t)
patient = {}
patient["UID"] = data.SOPInstanceUID
patient["Age"] = data.PatientAge
patient["Sex"] = data.PatientSex
patient["Modality"] = data.Modality
patient["BodyPart"] = data.BodyPartExamined
patient["ViewPosition"] = data.ViewPosition
patients.append(patient)
df_patients = pd.DataFrame(patients, columns=["UID", "Age", "Sex", "Modality", "BodyPart", "ViewPosition"])
df_patients["Age"] = pd.to_numeric(df_patients["Age"])
# Построим гистограмму
sorted_ages = np.sort(df_patients["Age"].values)
plt.style.use('seaborn-whitegrid')
plt.figure(figsize=(15, 5))
plt.hist(sorted_ages[:-2], bins=[i for i in range(100)])
plt.title("Распределение по возрасту", fontsize=18, pad=10)
plt.xlabel("возраст", labelpad=10)
plt.xticks([i*10 for i in range(11)])
plt.ylabel("Количество", labelpad=10)
plt.show()
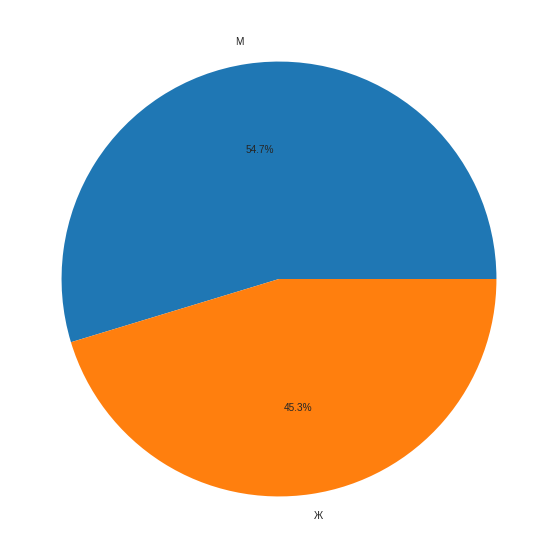
А теперь посмотрим на процентное соотношение мужчин и женщин.
plt.figure(figsize=(10, 10))
plt.pie([df_patients[df_patients["Sex"] == "M"].shape[0], df_patients[df_patients["Sex"] == "F"].shape[0]], labels=["М", "Ж"], autopct='%1.1f%%')
plt.show()
Подробно почитать про все метатеги тут.
Спасибо за внимательность Подправил
а можно ли из DICOM тега вынести данные по типу: компрессия (thikness) или лучевая нагрузка?
На этапе гистограмм возрастов появляется ошибка: UserWarning: Invalid value for VR UI: 'ID_fff6f183d'. Please see <https://dicom.nema.org/medical/dicom/current/output/html/part05.html#table_6.2-1> for allowed values for each VR. warnings.warn(msg)
По поводу thikness и лучевую нагрузку на практике не нужно было и не встречал*(
Ivan 17 Апрель 2021
В коде парсинга метаданных есть нехороший момент. Сначала в переменную data кладем список файлов, а затем в цикле читаем туда dicom.