Рассмотрим алгоритм сегментации на основе графов Felsenszwalb из библиотеки skimage. Алгоритм производит сегментацию многоканального (например, RGB) изображения с использованием быстрой кластеризации на основе минимального связующего дерева на сетке изображения. Параметр scale задает уровень наблюдения. Чем выше scale, тем меньше сегментов и они большего размера. sigma - это диаметр гауссова ядра, используемого для сглаживания изображения перед сегментацией.
Количество производимых сегментов, а также их размер можно контролировать только косвенно через scale. Размер сегмента в изображении может сильно различаться в зависимости от контраста. Для изображений RGB алгоритм использует евклидово расстояние между пикселями в цветовом пространстве.
Нашей задачей будет в уже знакомом нам рентген снимке попробовать сегментировать оба легких.
example = 'stage_2_images/ID_01fe90211.dcm'
imagedata= pydicom.dcmread(example)
# Импортируем нужные библиотеки
import numpy as np
from skimage.exposure import equalize_hist
from skimage.filters.rank import median
from skimage.morphology import disk
from skimage.segmentation import felzenszwalb
from skimage.transform import rescale
Занулим значения больше 65
im_thres = imagedata.pixel_array.copy()
im_thres[im_thres > 65] = 0
Масштабируем и фильтруем и нормализовываем картинку
im_small = rescale(im_thres, 0.5)
im_small_filt = median(im_small, disk(50))
im_small_filt = equalize_hist(im_small_filt)
Запускаем сам алгоритм
segments = felzenszwalb(im_small_filt, scale=0.5)
Визуализируем и посмотрим, что получилось
f = plt.figure(figsize=(12,12))
ax = f.add_subplot(121)
ax2 = f.add_subplot(122)
ax.imshow(imagedata.pixel_array, cmap=plt.cm.bone)
ax2.imshow(segments, cmap=plt.cm.bone)
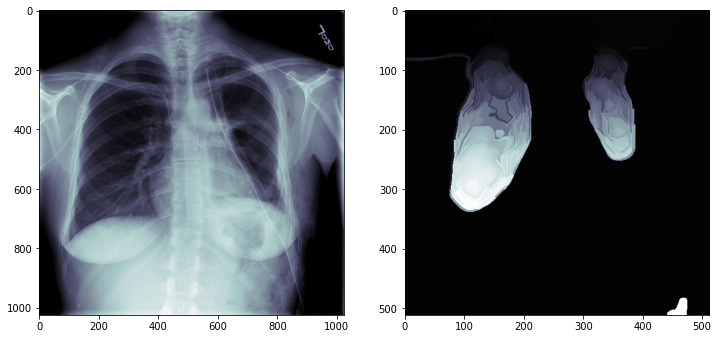
Получилось достаточно условно сегментировать легкие. Что бы получилось лучше нужно подобрать параметры.
Самостоятельная работа: подобрать параметры для лучшей сегментации легких.