Анализ медицинских изображений в Python: Исследование медицинских изображений / Загрузка и визуализация
Установим библиотеку nibabel.
pip install nibabel
Импортируем все необходимые для работы библиотеки
import nibabel # библиотека для работы с NIfTI-1 Data Format
import numpy as np # numpy для манипуляций с изображением
from matplotlib import cm # цветовые схемы для визуализации
from matplotlib import pyplot as plt # библиотека для визуализации
Считаем файл, используя метод load , а для получение изображения нужно использовать функцию get_fdata. Будем работать с файлом volume_pt5/volume-44.nii
filepath = 'volume_pt5/volume-44.nii'
imagedata = nibabel.load(filepath)
array = imagedata.get_fdata()
Дополнительно развернем изображение на 90 градусов и посмотрим на shape содержимого файла.
array = np.rot90(np.array(array))
print(array.shape)
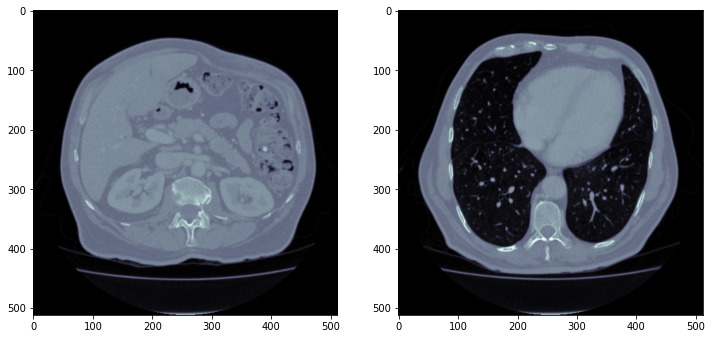
Результатом вывода будет Высота x Ширина x Глубина - (512, 512, 119). Глубина тут означает срезы КТ. Давайте визуализируем 50 и 118 срез КТ.
f = plt.figure(figsize=(12,12))
ax = f.add_subplot(121)
ax2 = f.add_subplot(122)
ax.imshow(array[...,50].astype(np.float32), cmap=plt.cm.bone)
ax2.imshow(array[...,118].astype(np.float32), cmap=plt.cm.bone)
Самостоятельная работа: визуализируйте другие срезы КТ.